(文/王贝贝) 2025年5月6日,中心李君一教授课题组22级硕士生王贝贝同学以第一作者在《Bioinformatics》期刊发表论文“MSNGO: multi-species protein function annotation based on 3D protein structure and network propagation”。《Bioinformatics》是Oxford Academic出版社旗下数学与计算生物学方向的JCR一区期刊,也是生物信息学领域的顶级期刊,影响因子(IF)为5.8。
图 1 模型MSNGO在线发表网页
王贝贝同学在李君一教授指导下,从本科生阶段开始接触生物信息学研究,对使用AI算法解决生物学问题有强烈的兴趣。她曾参与多项蛋白质功能预测的课题。在硕士期间,她专注于多物种蛋白质功能预测方法的研究,结合AlphaFold2预测的蛋白质三维结构,提出了一种创新性的多物种蛋白质功能注释框架——MSNGO。这一成果不仅解决了跨物种数据整合的难题,还显著提升了功能预测的准确性。此外,她在硕士期间的另一成果E-MSNGO也被CCF C类会议2025 International Conference on Intelligent Computing(ICIC2025)录用。
蛋白质功能预测是理解生命活动机制的重要工具,但传统的实验方法效率低、成本高,难以满足大规模数据分析的需求。近年来,随着AlphaFold2等高精度蛋白质结构预测工具的出现,基于结构的蛋白质功能预测方法取得了突破性进展。然而,现有的多物种预测方法仍面临数据整合困难、跨物种知识迁移不足等挑战,尤其是对于功能注释稀疏的物种,预测效果往往不尽如人意。
针对上述问题,论文提出了一种基于Alphafold2 3D蛋白质结构和网络传播的多物种蛋白质功能预测模型MSNGO。该模型通过整合蛋白质序列特征、结构特征以及蛋白质相互作用网络(PPI)和同源相似性网络,构建了一个异构网络,实现了跨物种的功能标签传播。MSNGO主要包含三个阶段:首先在特征提取阶段,MSNGO使用序列特征提取工具ESM-2提取了序列特征,将结构数据转化为氨基酸接触图,使用层次图卷积模型提取了结构特征,利用序列同源和PPI网络搭建了包含13个物种的异构网络。其次在训练阶段,MSNGO使用图注意力机制为异构网络学习有效权重,节点聚合了来自两种网络不同的信息,实现了多物种的网络传播。最后在预测阶段加入了训练集的标签传播,补充功能标签的直接信息传播。结果表明,MSNGO通过融合结构数据,使得预测性能有大幅提升,其在多物种和单物种的预测任务上均有出色的表现,并且性能显著优于现有的多物种蛋白质功能预测方法(如DeepGraphGO、SPROF-GO和PSPGO)。
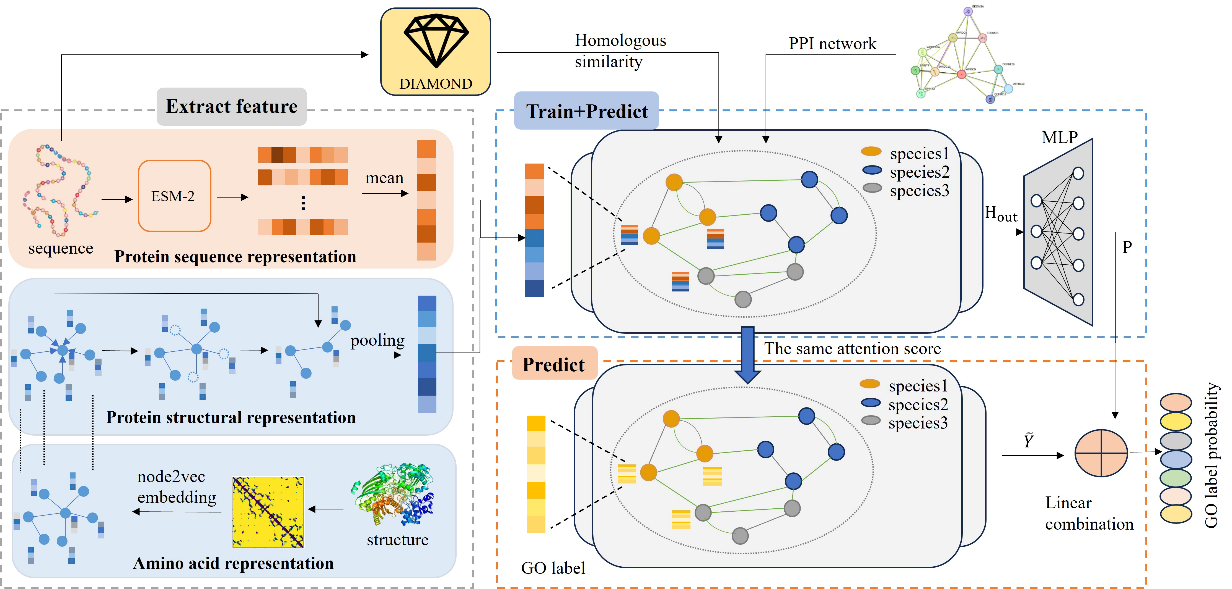
图 2 MSNGO模型示意图
论文第一作者为王贝贝同学,通讯作者为李君一教授。中心的王轩教授也参与了论文的研究工作。该研究得到了国家自然科学基金面上项目的资助。
(审核:李君一)

