(文/黄雨锐) 2025年4月12日,中心李君一教授课题组21级本科生黄雨锐和袁汝喆同学的关于合成致死基因预测的研究成果“Struct2SL: Synthetic lethality prediction based on AlphaFold2 structure information and Multilayer Perceptron”被《Computational and Structural Biotechnology Journal》期刊正式接收并在线发表。CSBJ期刊由Elsevier出版,是计算生物学和结构生物学领域的权威期刊,专注于发表计算方法、结构生物学以及生物技术相关领域的前沿研究成果,在JCR多个领域中被分类为Q1区期刊,其影响因子(IF)为4.5。
黄雨锐和袁汝喆同学在李君一老师指导的大一立项《基于python的癌症科普小爬虫及其可视化》中,对科研产生了极大的兴趣。他们在大二的时候加入了李君一老师课题组,同时进行大学生创新创业训练计划项目《基于深度学习的癌症合成致死基因预测》的研究。在此过程中他们积累了丰富的科研经验,提升了数据分析和模型构建能力,为后续的深入研究奠定了坚实基础。在大创项目结项以后,他们继续进行基于AlphaFold2蛋白质结构特征和深度学习的合成致死基因预测模型的研究。大四毕业之际,研究结果终于付梓。
合成致死(Synthetic Lethality, SL)是癌症治疗中的一个重要概念,指两个基因单独出现异常不会导致细胞死亡,但同时出现异常则会导致细胞死亡的现象。这一特性为癌症治疗提供了特异性靶点,有望在不影响正常细胞的情况下杀死癌细胞。然而,传统的实验方法在识别合成致死基因对时面临成本高、耗时长等挑战,限制了其广泛应用。因此,开发高效的计算方法来预测合成致死基因对具有重要的科学意义和应用价值。
近年来,基于深度学习的计算方法在合成致死基因对预测领域取得了显著进展,但现有方法大多仅依赖于基因表达或蛋白质相互作用网络的拓扑结构信息,忽略了蛋白质的三维结构特征以及蛋白质序列信息,而这些信息对于理解基因功能和相互作用至关重要。针对现有方法的局限性,本研究提出了一种基于蛋白质结构信息和多层感知器的合成致死基因对预测框架——Struct2SL。该框架的创新之处在于通过整合蛋白质序列、蛋白质相互作用(PPI)网络和蛋白质三维结构信息,构建基因相互作用的特征表示,从而实现对合成致死基因对的高精度预测。
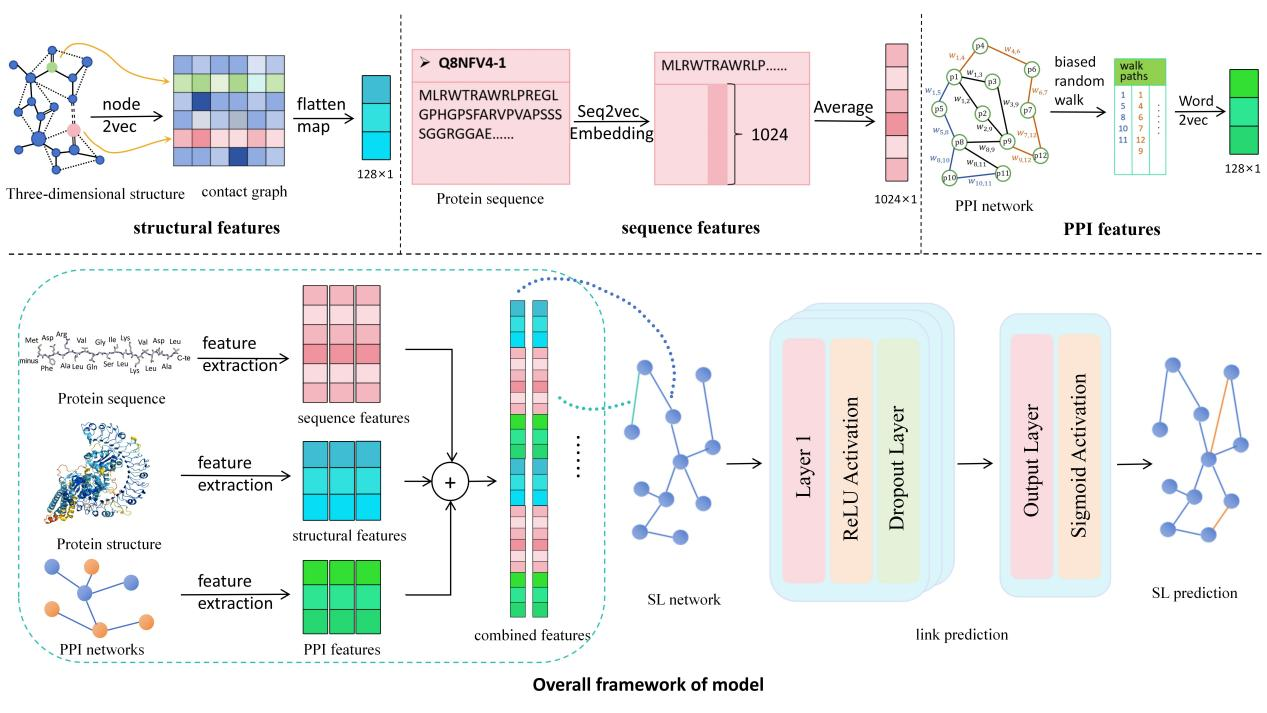
图 1 Struct2SL模型示意图
Struct2SL框架包含四个关键阶段:首先,利用AlphaFold2预测蛋白质的三维结构,并提取蛋白质的序列特征和PPI网络特征,这些特征为后续的基因嵌入提供了基础信息。接着,通过蛋白质与基因的映射关系,将蛋白质的特征信息整合到基因层面,生成基因的初步嵌入表示,使得基因的特征表示更加丰富和准确。然后,基于已知的合成致死基因对构建合成致死图,进一步优化基因的嵌入表示。最后利用多层感知器对基因对的合成致死性进行预测。将Struct2SL模型与现有的4种先进方法进行了比较,使用AUC、AUPR和F1-score指标评估了模型性能。结果表明,Struct2SL在所有评估指标上均优于其他方法。此外,论文还通过分析蛋白质序列、结构和PPI网络特征对预测结果的贡献,以案例分析的形式讨论了模型的预测准确度和泛化性能。
该研究不仅为合成致死基因对的预测提供了一种新的高效计算方法,还开发了一个名为“Struct2SL”的合成致死基因查询服务器,为研究人员提供了一个便捷的工具,用于查询和预测合成致死基因对。
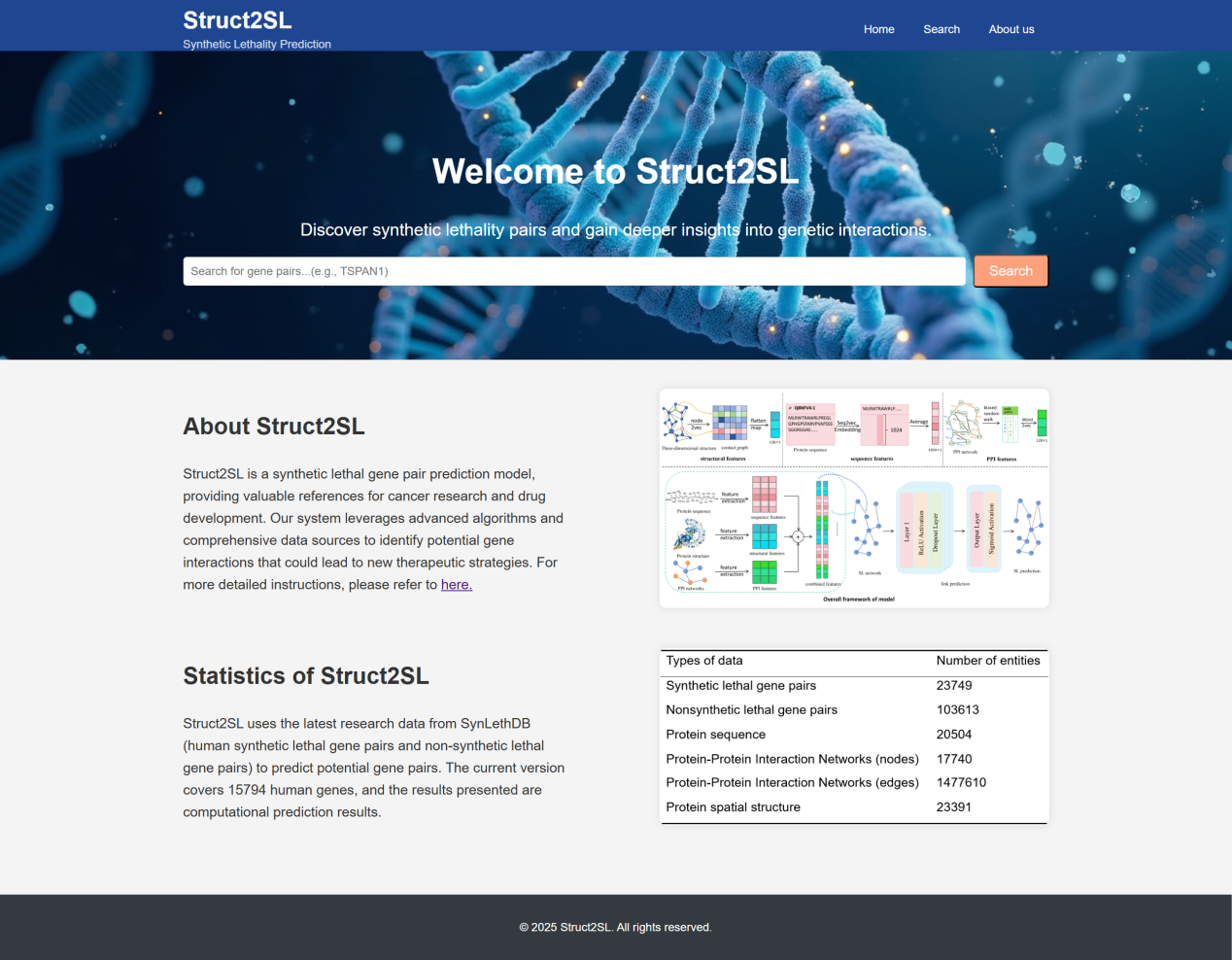
图 2 合成致死基因查询主页
论文共同第一作者为21级本科生黄雨锐和袁汝喆同学,通讯作者为李君一教授。中心20级本科生李亚轩同学和23级硕士研究生邢哲铭同学参与了论文的研究工作。

