(焦沛舜/文、图)2023年10月,中心李君一副教授课题组的论文“Struct2GO:protein function prediction based on Graph pooling algorithm and AlphaFold2 structure information”被《Bioinformatics》期刊正式接收并在线发表。《Bioinformatics》是Oxford Academic出版社旗下数学与计算生物学方向的JCR一区期刊,也是生物信息学领域的顶级期刊,影响因子为5.8。
李君一课题组长期进行蛋白质功能预测相关研究,王立新等人提出的Prot2GO模型基于蛋白质的序列和PPI数据有效地标注GO标签。张晓帅等人设计基于注意力机制的异构网络链接预测模型HNetGO进行蛋白质功能预测。吴凯涛等人结合图神经网络和标签传播算法创新地提出了多物种蛋白质功能预测模型PSPGO。
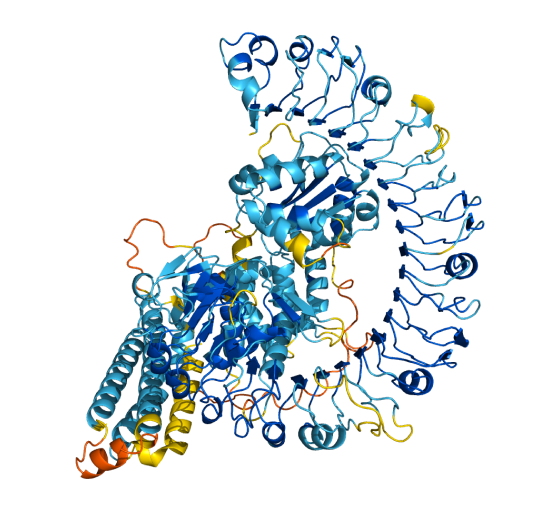
图片来源:AlphaFold2官方网站(https://alphafold.ebi.ac.uk/)
图1 Alphafold2预测植物抗病蛋白(Q8W3K0)结构
近年来,蛋白质结构预测有了显著突破,DeepMind团队的AlphaFold2模型将蛋白质结构预测的精度提高到了原子水平。目前,基于深度学习的蛋白质功能预测模型通常是从蛋白质序列中提取特征,并将其与蛋白质-蛋白质相互作用网络相结合,以获得较好的结果。然而,对于未在蛋白-蛋白相互作用网络中的新测序蛋白,这种模型无法进行有效的预测。针对这一问题,该论文提出了结合蛋白质结构和序列数据的Struct2GO模型,提高了蛋白质功能预测的精度和模型的通用性。
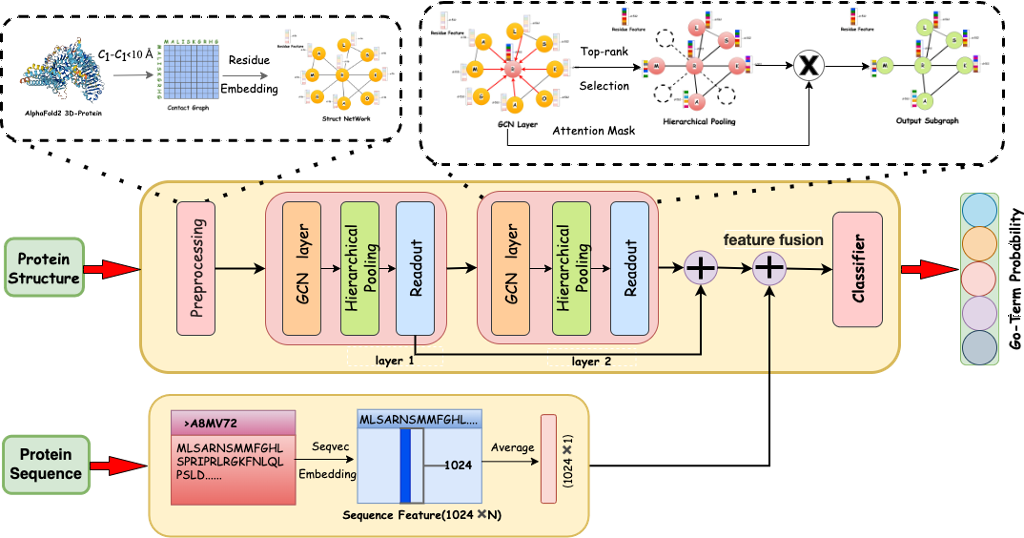
图2 Struct2GO算法框架
该论文提出了一种基于多源数据融合的蛋白质功能预测模型Struct2GO。该模型以蛋白质序列信息和蛋白质结构信息为输入,通过预训练模型SeqVec提取序列特征,通过基于自关注机制的分层图池化模型提取结构特征。为了最大限度地利用AlphaFold2提供的蛋白质结构信息,通过Node2vec在蛋白质结构网络中预训练残差级嵌入,然后将其作为池化模型的初始节点特征。大量实验表明,结合结构和序列的蛋白质功能预测模型可以显著提高预测精度。同时,该模型消除了PPI网络对特征提取的限制,显著提高了模型的泛化能力。
论文第一作者为21级硕士研究生焦沛舜,通讯作者为李君一副教授。中心王轩教授和22级硕士研究生王贝贝共同参与论文研究工作。(审核 刘洋)

