(曹源、刘洋/文、图)2023年2月,中心李君一副教授课题组的论文“SLGNN: synthetic lethality prediction in human cancers based on factor-aware knowledge graph neural network”被《Bioinformatics》期刊正式接收并在线发表。《Bioinformatics》是Oxford Academic出版社旗下数学与计算生物学方向的中科院一区期刊,也是生物信息学领域的顶级期刊,影响因子为6.937。

图1 Bioinformatics期刊
合成致死(Synthetic Lethality,SL)是一种基因之间的相互作用,这种相互作用表现为当两个或多个基因同时发生突变时会导致细胞死亡,而只有其中一个基因发生突变时则不会产生致死效果。肿瘤细胞的产生通常伴随着特异的基因失活,使用特定的药物抑制该基因的 合成致死基因,就可以在杀死肿瘤细胞的同时,不损伤正常的健康细胞。基于合成致死策略,可以避免细胞毒性治疗的缺陷,从而实现个性化的癌症治疗方式,这为抗癌药物的研究提供了新方向。相较于实验方法,基于计算的合成致死预测方法可以通过先筛查后验证的方式,有效地缩短抗癌靶向药物的研发周期,同时避免高通量测序技术所带来的高研发成本。目前基于网络表征的合成致死预测方法基本上遵循端到端的深度学习模式,其性能良好但缺乏可解释性,这种不可解释性使得预测结果很难发展到生物实验或临床试验。
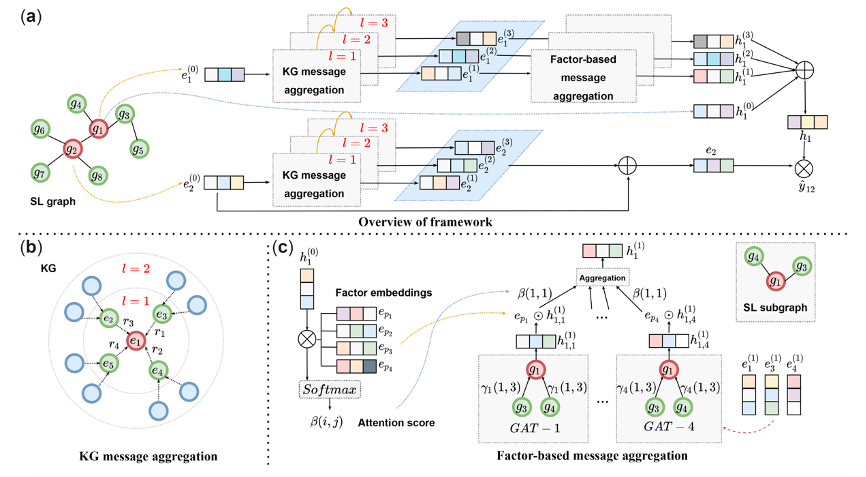
图2 SLGNN算法框架
该论文提出了一种基于因素感知的合成致死预测算法,通过将生物医药知识图谱中表示各种生物过程的关系建模为导致合成致死的因素,并将这些因素引入模型训练过程的方式来提升模型的表达能力,同时提高模型的可解释性。论文提出一个基本假设,即不同的生物过程导致了基因之间的合成致死相互作用,这些生物过程可能是基因之间共同表达、基因之间的共突变或基因共同参与某一代谢途径等。将导致合成致死的因素建模为知识图谱中所有关系的不同加权组合,同时假设这些因素对于不同的合成致死相互作用具有不同的重要性,通过分析网络卷积过程中因素的注意力系数,以此对模型的预测结果进行解释。同一数据集下的实验证实,该方法的预测效果相比于目前合成致死预测的基准方法有了明显提升,同时具有更好的可解释性。
论文第一作者为20级硕士研究生朱焱,通讯作者为李君一副教授。中心王轩教授、刘洋助理教授以及21级硕士研究生周雨桓共同参与论文研究工作。(审核 刘洋)

